
Enregistrez les noms des espèces et les valeurs des coefficients stœchiométriques dans des variables
nom_A="peroxodisulfate "
nom_B="iodure"
nom_C="diiode"
nom_D="ion sulfate"
coef_A=1
coef_B=2
coef_C=1
coef_D=2
print("le nom de l'espèce A est :",nom_A)
print("le coefficient stoechiométrique de l'espèce A est ",coef_A)
On peut enregistrer ces valeurs de façon plus interactive avec la fonction input()
nom_A=input("nom de l'espèce A ? ")
nom_B=input("nom de l'espèce B ? ")
nom_C=input("nom de l'espèce C ? ")
nom_D=input("nom de l'espèce D ? ")
coef_A=float(input("coefficient stoechiométrique de A ? "))
coef_B=float(input("coefficient stoechiométrique de B ? "))
coef_C=float(input("coefficient stoechiométrique de C ? "))
coef_D=float(input("coefficient stoechiométrique de D ? "))
print("le nom de l'espèce A est :",nom_A)
print("le coefficient stoechiométrique de l'espèce A est ",coef_A)
Le milieu réactionnel est constitué d’un mélange de deux solutions aqueuses.5mL de peroxodisulfate à 0.025mol/L et 25mL d’ions iodure à 0.005 mol/L. Déterminez les quantités de matière initiales
VA=5E-3 CA=0.025 VB=25E-3 CB=5E-3 NAi=CA*VA NBi=CB*VB NCi=0 NDi=0 print(NAi) print(NBi)
Calculez xmax et donnez le réactif limitant
nom_A="peroxodisulfate "
nom_B="iodure"
nom_C="diiode"
nom_D="ion sulfate"
coef_A=1
coef_B=2
coef_C=1
coef_D=2
VA=5E-3
CA=0.025
VB=25E-3
CB=5E-3
NAi=CA*VA
NBi=CB*VB
NCi=0
NDi=0
#if(abs(NAi/coef_A-NBi/coef_B)<1E-9):
if(NAi/coef_A == NBi/coef_B):
xmax=NAi/coef_A
print("le mélange est stoechiométrique")
elif (NAi/coef_A>NBi/coef_B):
print("B est le réactif limitant")
xmax=NBi/coef_B
else:
print("A est le réactif limitant")
xmax=NAi/coef_A
print("xmax = ",xmax)
Représentons l’état initial à l’aide d’un histogramme
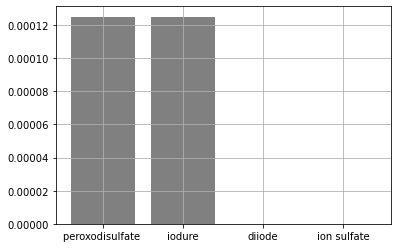
#py003
import matplotlib.pyplot as plt
nom_A="peroxodisulfate "
nom_B="iodure"
nom_C="diiode"
nom_D="ion sulfate"
coef_A=1
coef_B=2
coef_C=1
coef_D=2
espece= [nom_A, nom_B, nom_C,nom_D]
VA=5E-3
CA=0.025
VB=25E-3
CB=5E-3
NAi=CA*VA
NBi=CB*VB
NCi=0
NDi=0
quantite = [NAi,NBi,NCi,NDi]
bars=plt.bar(espece, quantite,color='grey' )
bars[2].set_facecolor('gold')
plt.grid()
plt.show()
Représentons l’état final à l’aide d’un histogramme
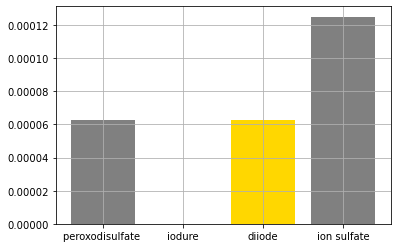
import matplotlib.pyplot as plt
nom_A="peroxodisulfate "
nom_B="iodure"
nom_C="diiode"
nom_D="ion sulfate"
espece= [nom_A, nom_B, nom_C,nom_D]
coef_A=1
coef_B=2
coef_C=1
coef_D=2
VA=5E-3
CA=0.025
VB=25E-3
CB=5E-3
NAi=CA*VA
NBi=CB*VB
NCi=0
NDi=0
xmax =min(NAi/coef_A,NBi/coef_B)
NAf=NAi-coef_A*xmax
NBf=NBi-coef_B*xmax
NCf=NCi+coef_C*xmax
NDf=NDi+coef_D*xmax
quantite = [NAf,NBf,NCf,NDf]
bars=plt.bar(espece, quantite,color='grey' )
bars[2].set_facecolor('gold')
print(xmax)
plt.grid()
plt.show()
Représentons l’évolution des quantités de matière en fonction de l’avancement.
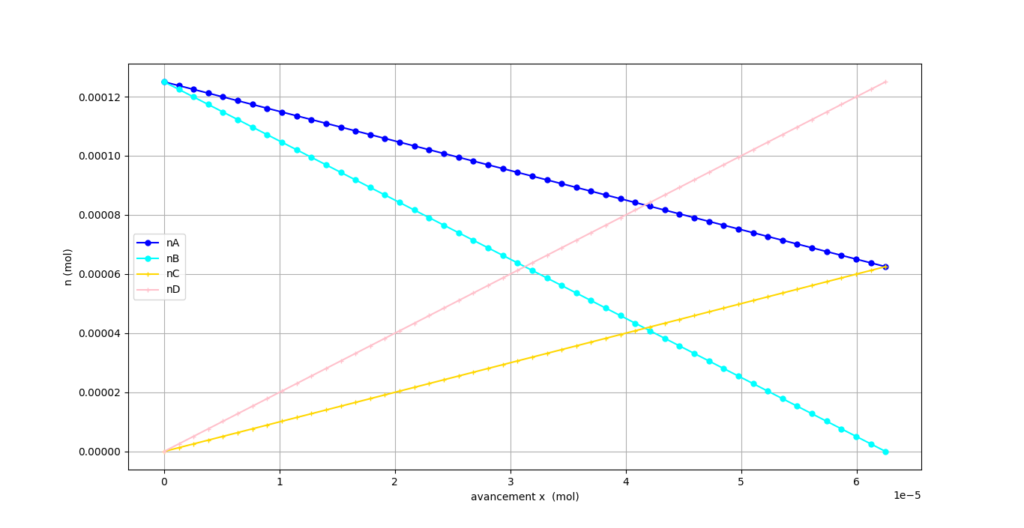
import matplotlib.pyplot as plt
coef_A=1
coef_B=2
coef_C=1
coef_D=2
VA=5E-3
CA=0.025
VB=25E-3
CB=5E-3
NAi=CA*VA
NBi=CB*VB
NCi=0
NDi=0
x=[]
na=[]
nb=[]
nc=[]
nd=[]
xmax =min(NAi/coef_A,NBi/coef_B)
dx=xmax/50
xi=0
while xi<xmax:
x.append(xi)
na.append(NAi-coef_A*xi)
nb.append(NBi-coef_B*xi)
nc.append(NCi+coef_C*xi)
nd.append(NDi+coef_D*xi)
xi=xi+dx
plt.plot(x,na,'blue',label='nA',marker='o',markersize=5)
plt.plot(x,nb,'cyan',label='nB',marker='o',markersize=5)
plt.plot(x,nc,'gold',label='nC',marker='o',markersize=5)
plt.plot(x,nd,'pink',label='nD',marker='o',markersize=5)
plt.legend()
plt.ylabel('n (mol)')
plt.xlabel('avancement x (mol)')
plt.grid()
plt.show()
remplaçons les listes par des tableaux numpy
import matplotlib.pyplot as plt
import numpy as np
coef_A=1
coef_B=2
coef_C=1
coef_D=2
VA=5E-3
CA=0.025
VB=25E-3
CB=5E-3
NAi=CA*VA
NBi=CB*VB
NCi=0
NDi=0
xmax =min(NAi/coef_A,NBi/coef_B)
x=np.linspace(0,xmax,50)
na=NAi-coef_A*x
nb=NBi-coef_B*x
nc=NCi+coef_C*x
nd=NDi+coef_D*x
plt.plot(x,na,'blue',label='nA',marker='o',markersize=5)
plt.plot(x,nb,'cyan',label='nB',marker='o',markersize=5)
plt.plot(x,nc,'gold',label='nC',marker='+',markersize=5)
plt.plot(x,nd,'pink',label='nD',marker='+',markersize=5)
plt.legend()
plt.ylabel('n (mol)')
plt.xlabel('avancement x (mol)')
plt.grid()
plt.show()
